RやPythonで変数間の相関を見るために、何気なく使う相関行列
実はとても奥深いことを知りました。
今回はRとPythonの算出方法の違いについても触れていきます。(pythonを使ったのはいつぶりだろうか...)
データセットの作成
まずはデータセットを作成します。データは6人の小学生の身長、体重、年齢、50m走のタイムとしましょう。
Rの場合
df <- data_frame(heights=c(110,120,130,130,150,155), weight = c(25,25,25,35,45,50), age = c(6,7,9,10,10,12), time50m =c(15,11,10,10,9,9) )
Pythonの場合
import numpy as np import pandas as pd df=pd.DataFrame(np.array([[110,120,130,130,150,155], [25,25,25,35,45,50], [6,7,9,10,10,12], [15,11,10,10,9,9]]).T,columns=["height","weight","age","time50m"])
| heights | weight | age | time50m |
|---|---|---|---|
| 110 | 25 | 6 | 15 |
| 120 | 25 | 7 | 11 |
| 130 | 25 | 9 | 10 |
| 130 | 35 | 10 | 10 |
| 150 | 45 | 10 | 9 |
| 155 | 50 | 12 | 9 |
相関行列の算出
続いて、RとPythonそれぞれで相関行列を算出してみます。
Rの場合
> cor(df) heights weight age time50m heights 1.0000000 0.9235329 0.9261982 -0.8499811 weight 0.9235329 1.0000000 0.8601935 -0.6511951 age 0.9261982 0.8601935 1.0000000 -0.8516625 time50m -0.8499811 -0.6511951 -0.8516625 1.0000000
Pythonの場合
df.corr()
| height | weight | age | time50m | |
|---|---|---|---|---|
| height | 1.000000 | 0.923533 | 0.926198 | -0.849981 |
| weight | 0.923533 | 1.000000 | 0.860194 | -0.651195 |
| age | 0.926198 | 0.860194 | 1.000000 | -0.851662 |
| time50m | -0.849981 | -0.651195 | -0.851662 | 1.000000 |
身長、体重、年齢はそれぞれ正の相関、50mタイムは負の相関です。これは、直感的に分かりやすいですね。 RもPythonも結果は同じになりました
それでは、データに欠損を作ってみましょう。
Rの場合
df$weight[c(5,6)] <- NA df$time50m[1] <- NA
Pythonの場合
df.loc[[4,5],"weight"] = np.nan df.loc[0,"time50m"] = np.nan
| heights | weight | age | time50m |
|---|---|---|---|
| 110 | 25 | 6 | NA |
| 120 | 25 | 7 | 11 |
| 130 | 25 | 9 | 10 |
| 130 | 35 | 10 | 10 |
| 150 | NA | 10 | 9 |
| 155 | NA | 12 | 9 |
これで相関行列を出してみると
R
> cor(df) heights weight age time50m heights 1.0000000 NA 0.9574271 NA weight NA 1 NA NA age 0.9574271 NA 1.0000000 NA time50m NA NA NA 1
Python
df.corr()
| height | weight | age | time50m | |
|---|---|---|---|---|
| height | 1.000000 | 0.923533 | 0.926198 | -0.849981 |
| weight | 0.923533 | 1.000000 | 0.860194 | -0.651195 |
| age | 0.926198 | 0.860194 | 1.000000 | -0.851662 |
| time50m | -0.849981 | -0.651195 | -0.851662 | 1.000000 |
おや、どうやら結果が違うようです。
Rのcor関数について
これは、欠損値の扱い方がRとPythonでは違うために起こります。Rでは欠損を除いて関数を適応する際にna.rm=Tと指定することがあるかと思います。(meanなど)
しかし、cor関数にはna.rmが指定できず、代わりにuseという引数を使って、欠損の扱いを指定できます。
useには
- everything(デフォルト)
- all.obs
- complete.obs
- pairwise.complete.obs
が指定できます。
試しにやってみましょう。
R
>#everthing > cor(df,use = "everything") heights weight age time50m heights 1.0000000 NA 0.9261982 NA weight NA 1 NA NA age 0.9261982 NA 1.0000000 NA time50m NA NA NA 1 > > #all.obs > cor(df,use = "all.obs") cor(df, use = "all.obs") でエラー: cov/cor 関数に欠損した観測値があります > > #complete.obs > cor(df,use = "complete.obs") heights weight age time50m heights 1.0000000 0.5000000 0.9449112 -1.0000000 weight 0.5000000 1.0000000 0.7559289 -0.5000000 age 0.9449112 0.7559289 1.0000000 -0.9449112 time50m -1.0000000 -0.5000000 -0.9449112 1.0000000 > > #pairwise.complete.obs > cor(df,use="pairwise.complete.obs") heights weight age time50m heights 1.0000000 0.5222330 0.9261982 -0.9669876 weight 0.5222330 1.0000000 0.7302967 -0.5000000 age 0.9261982 0.7302967 1.0000000 -0.8882348 time50m -0.9669876 -0.5000000 -0.8882348 1.0000000
順番にいきましょう。まず、デフォルトのeverythingですが、これは変数に欠損が一つでもあれば、その変数とその他の変数との相関係数を算出しません。
all.obsはさらに厳しくて、全てのデータが完璧に揃っていた場合のみ相関行列を算出します。
complete.obsをpairwise.complete.obsが少しややこしいですが、下の図をご覧ください。
complete.obsは、欠損のある行を全て除いて相関係数を算出しているのに対して、pairwise.complete.obsは欠損のある変数との相関係数のみcomplete.obsを使って相関係数を算出しています。
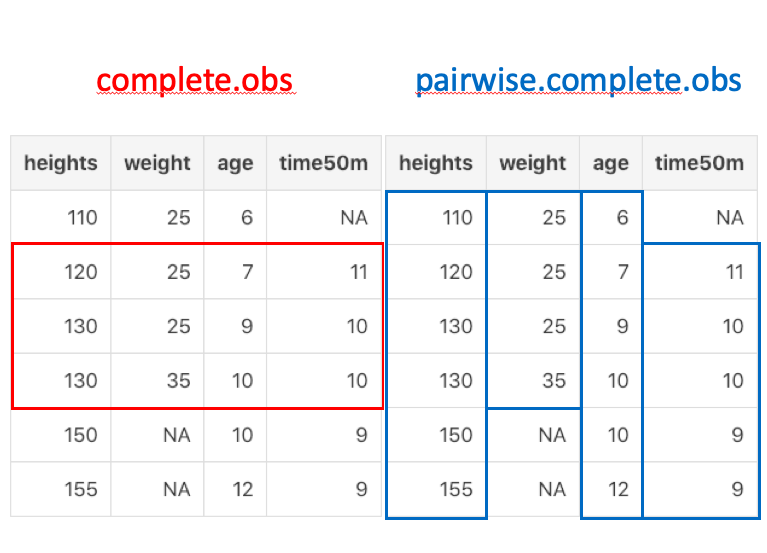
例えば、weightとtime50mの時は真ん中3人分だけが使われているため、complete.obsと同じ結果になっています。逆にheightとageは欠損がないので、everytingと同様の結果になっています。
Pythonの場合はpairwise.complete.obsと同じことを行なっています。
ちなみにRでは共分散を算出するcov関数でも同様の引数を指定できます!
相関行列の注意点
ここでpairwise.complete.obsの相関係数、もしくはpythonの相関係数に注目してみてください。本来、0.92と高い相関にあったheightとweightの相関係数が0.52と小さな相関になっています。 これは本来相関関係にあるはずのペアデータが欠損しているためです。
大量のデータかつ完全ランダムな欠損であれば、pairwise.complete.obsやpythonのデフォルトでも問題ありませんが、欠損に規則性があった場合、(例えば高学年は体重を測定していないとか)重要な相関関係を見逃す可能性もあります。
普段何気なく使っている相関行列も欠損がどのように処理をされているか知っておいて損はないですね!